Tras el descanso navideño vuelvo a la carga con una nueva publicación. En un esfuerzo por intentar que no sea una cosa muy monotema hoy traigo una publicación que no es exactamente de cáncer. Se trata de un algoritmo desarrollado Google Brain Team de Google para identificar mutaciones en genomas. Como esto se está haciendo hoy en día en miles de genomas de cáncer, es una publicación que encaja perfectamente en la temática.
12/01/2018
DeepVariant: Highly Accurate Genomes With Deep Neural Networks
Otro link más detallado: https://www.biorxiv.org/content/early/2016/12/21/092890
Introducción
Para los que no estén muy puestos en el tema, se trata del uso de las conocidas como Neural Networks para la identificación de mutaciones en el genoma. Hoy en día es una técnica que se usa no solo para este propósito, sino para infinidad de ellos. Como no es el objetivo concreto de este post el explicarlas, os dejo un video que da una idea general de como funcionan para otro caso diferente: proponer las sugerencias de videos en Youtube:
Ahora que ya sabemos un poco de que va esto de las redes neuronales y que parecen ser especialmente buenas realizando tareas de identificar cosas si disponemos de ejemplos suficientes para entrenarlas, ¿porque no las usamos para analizar nuestro genoma? Pues dicho y hecho, es lo que el grupo de trabajo de Google para el desarrollo de esta tecnología acaban de publicar.
La herramienta
Entonces, ¿Le han metido muchos ejemplos, han evaluado las salidas y han visto como de bien lo hacen? Pues no exactamente, el esquema de como trabaja el programa se puede ver en el diagrama siguiente:
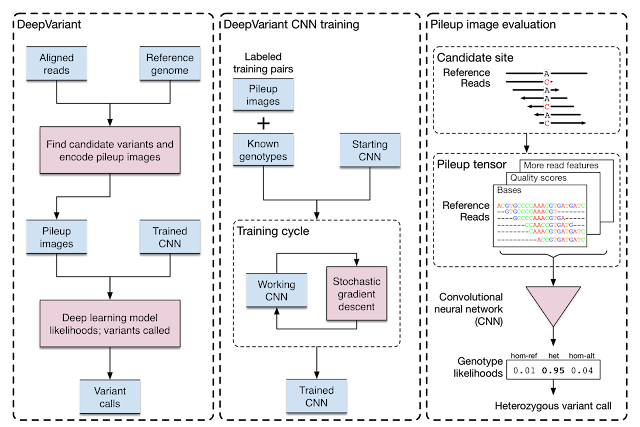
La gracia de la solución que aportan es que el programa no evalúa exactamente secuencias de DNA, sino la imágenes alineadas de los miles de millones de fragmentos de DNA que las máquinas de secuenciación generan, lo que ellos llaman el Pileup, que vendría a ser el apilamiento de estos reads (fragmentos secuenciados de DNA). De hecho pileup format es desde hace muchos años un formato utilizado para representar estos alineamiento.
Las neural networks simulan en este caso el comportamiento exacto del investigador sentado frente a su ordenador, observando una región concreta del genoma y decidiendo si lo que ve frente a sus ojos se trata de una mutación o no. Imágenes como esta podemos generar millones para 1 solo paciente de cáncer, y esta evaluación visual por parte del investigador sólo se hace en unas pocas regiones en casos muy concretos: mirar una región conflictiva, evaluar errores, etc. Delegamos en otros algoritmos (Mutect, ASCAT, Pindel, Breakdancer, etc) el encontrar dichas mutaciones basándonos en otras propiedades más "matemáticas" de las mismas. Pero claro ¿que le supone a un ordenador evaluar unos cientos de millones de imágenes? Si sabe lo que busca y la red neuronal está ya construida, la verdad es que no más que un puñado de minutos.

Han evaluado la precisión con la que la red realiza esta tarea y en precisionFDA Truth Challenge han sido el método que mejor ha sabido capturar las mutaciones puntuales (SNV o SNP) y han ocupado un lugar destacado en las otras categorías al compararse con decenas de otras aplicaciones actuales destinadas exclusivamente a este cometido (para los curiosos son el la organización "Verily Life Sciences", ya que no encontrareis Google por ningún lado.
Como es natural en la visión empresarial de Google, invitan a todos los investigadores a usar su herramienta en su plataforma en la nube Google Cloud Platform (GCP).
Opinión
Que alguien con tan pocos conocimientos de redes neuronales de su opinión sobre ellas, debería estar prohibido en este subforo, por lo que más que opiniones divagaré un poco y dejaré que si alguno le apetece me discuta o contradiga.
Más allá de lo sorprendente de la estrategia, evaluar imágenes en vez de ponerse a recorrer texto como un loco, hay que reconocer que los resultados son sorprendentes como ya nos tienen acostumbrados las redes neuronales. Con el suficiente número de ejemplos y sabiendo qué información necesitan para entrenarse correctamente, acaban haciéndose las amas de todo proceso de selección, y en cáncer tenemos ejemplos para dar y regalar.
Pero creo que su fuerza es a su vez su debilidad. Ya que las neural networks entrenar a base de entrada-salida evaluando como de bien lo realizan y ajustándose en función a ello, pero ¿realmente conocemos en este caso la salida? No estamos hablando de algo que podamos evaluar a ciencia cierta como es "si el usuario hace finalmente click en el video sugerido" o "si efectivamente en la imagen sale una jirafa". Aquí se trata de algo diferente, se trata de encontrar lo desconocido.
Sabemos que hay muchas mutaciones desconocidas, de hecho los algoritmos para detectar mutaciones han sufrido enormes mejoras encontrando nuevas mutaciones, no solo siendo más eficientes en encontrar las que ya conocíamos. Me refiero a mutaciones en regiones de baja complejidad, flanqueadas por secuencias repetitivas, multiplicidad de una misma región en centenares o miles de posiciones diferentes del genoma, etc, etc.
¿Realmente buscamos ser un poco más precisos en encontrar lo que ya encontramos? A fin de cuentas este método obtiene un 99.9587% donde otros ya existentes sacan un 99.9%. ¿O necesitamos ser capaces de sumergirnos en ese oscuro mundo de las que actualmente no somos capaces de identificar? Aquí no se pueden dar datos aproximados porque como no lo sabemos...
Tengo la impresión que en este caso concreto (identificar mutaciones), las neural networks pueden ofrecer una solución rápida, eficiente y precisa a las tareas que ya estamos realizando, algo terriblemente útil para realizar otras investigaciones posteriores. Pero siguen sin solucionar el problema de identificar aquello que todavía se nos resiste en cáncer.








 , pero así de forma rápida decir que la estrategia se centra en minimizar una función mediante el método de Simplex Minimization de Nelder y Mead. La función que usa tiene en cuenta una frecuencia de aparición de mutaciones, una fitness o ventaja asignada a cada mutación y un parámetro de tránsito de células entrando y saliendo del sistema. De esta forma y limitando los valores que pueden albergar las diferentes variables en juego, se intenta encontrar la combinación que mejor explica las observaciones.
, pero así de forma rápida decir que la estrategia se centra en minimizar una función mediante el método de Simplex Minimization de Nelder y Mead. La función que usa tiene en cuenta una frecuencia de aparición de mutaciones, una fitness o ventaja asignada a cada mutación y un parámetro de tránsito de células entrando y saliendo del sistema. De esta forma y limitando los valores que pueden albergar las diferentes variables en juego, se intenta encontrar la combinación que mejor explica las observaciones.



 , que transforme el alcohol en aldehídos y posteriormente la enzima aldehido deshidrogenasa (ALDH2) que nos libra finalmente de los problemas con el alcohol, al menos aquellos asociados al daño de nuestro DNA.
, que transforme el alcohol en aldehídos y posteriormente la enzima aldehido deshidrogenasa (ALDH2) que nos libra finalmente de los problemas con el alcohol, al menos aquellos asociados al daño de nuestro DNA.

